【中英文題目】
An evaluation of the PacBio RS platform for sequencing and de novo assembly of a chloroplast genome
通過(guò)對葉綠體基因組的測序及從頭組裝對PacBio RS測序平臺進(jìn)行評估
【基本信息】
期刊:BMC GENOMICS
IF:4.276
年份:2013
【摘要】
背景:二代測序技術(shù)已經(jīng)可以為越來(lái)越多的非模式生物在全基因組水平上描述序列特征���,但是�����,測序讀長(cháng)較短���,基因覆蓋區具有偏向性����,后期組裝繁瑣��。PacBio RS測序平臺增加了reads長(cháng)度���,基因覆蓋區無(wú)偏向性���,因此���,最終產(chǎn)生的基因組序列就擁有較少的gap和較長(cháng)的contig����。但是�����,三代測序的缺點(diǎn)是成本和錯誤率都較高����。本次研究通過(guò)對Potentilla micrantha(委陵菜甘菊)葉綠體基因組的測序以及從頭組裝從而對PacBio RS測序平臺進(jìn)行評價(jià)�����。
結果:從葉綠體基因組中一共得到28,638個(gè)PacBio RS reads�,每個(gè)reads的平均長(cháng)度為1,902bp, 測序深度為320×��。對于單個(gè)contig��,PacBio RS測序數據完全覆蓋了葉綠體基因組的154,959bp (100% coverage)����,相比Illumina七個(gè)contig(90.59% coverage)����,而且��,對于GC富集區域也并沒(méi)有明顯偏好性�。后期序列的組裝與Illumina類(lèi)似�,允許在兩端的反向重復區域存在一些差異�。
結論:本次研究是基于葉綠體基因組PacBio測序數據進(jìn)行從頭組裝的第一次報道��,用來(lái)組裝的PacBio數據只產(chǎn)生一個(gè)較大的contig���,與Illumina相比����,產(chǎn)生的reads較長(cháng)并且具有較低的GC偏好性���。研究表明����,PacBio測序對于基因組研究具有很大的實(shí)用性����,相比Illumina產(chǎn)生的短reads���,它并不會(huì )產(chǎn)生很多gap和contig.
【研究思路】
取材:
Potentilla micrantha(以下簡(jiǎn)稱(chēng)P. micrantha)取自塞爾維亞的阿瓦拉山��,將其帶回實(shí)驗室并在適宜條件下培養����,促進(jìn)其快速生長(cháng)�,之后�,提取葉片DNA.
文庫構建:
分別利用PacBio RS和Illumina HiSeq2000對提取到的DNA進(jìn)行建庫
測序策略:
Pacific Biosciences PacBio RS利用單分子實(shí)時(shí)(SMRT)測序技術(shù)?����;HiSeq?測序系統既有Illumina和Solexa在邊合成邊測序上的優(yōu)勢�����,又融合了最新的光學(xué)圖譜��。
 信息分析:
信息分析:
【研究結果】
1.Illumina HiSeq2000和PacBio?RS的測序數據
在進(jìn)行誤差糾正之前�,提取只包含葉綠體基因組的序列�����,PacBio RS reads的平均長(cháng)度是3,936.66 bp, 一共含有223,483,907個(gè)核苷酸��,經(jīng)過(guò)HGAP誤差糾正之后���,還有28,638個(gè)PacBio?RS reads平均長(cháng)度為1,902.75 bp且一共含有54,492,250 bp核苷酸�����。經(jīng)過(guò)修剪以后����,Illumina reads一共含有7,164,496對�����,平均長(cháng)度在99.22bp�����,一共含有核苷酸1,421,726,349個(gè)�。
2.葉綠體基因組的組裝
PacBio RS與Illumina HiSeq2000所產(chǎn)生的數據以及序列的組裝的比較見(jiàn)圖1與表1
表1 P. micrantha葉綠體基因組的序列的統計
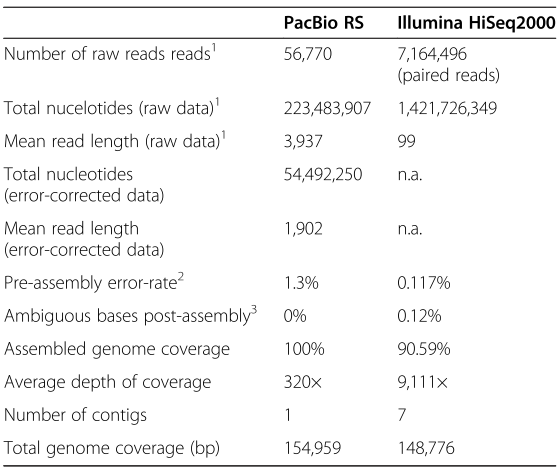
使用PacBio RS和Illumina HiSeq2000得到的P. Micrantha葉綠體基因組的測序數據匯總統計
1表示修建的Illumina reads
2表示誤差修正的PacBio reads和原始的Illumina reads
3表示與葉綠體一致性序列的比較

圖1?P. micrantha葉綠體基因組的序列覆蓋范圍?示意圖顯示的是將P. micrantha葉綠體基因組通過(guò)ABySS和Celera?assembler組裝得到的Illumina(黑色)和PacBio(綠色)contigs. 示意圖最上面的紅色的線(xiàn)條表示的是葉綠體基因�����,藍色加粗的區域表示的是基因組中反向重復的區域�����,Illumina和PacBio組裝得到的contig 1中并非IR唯一的部分用紅色表示出來(lái)
?3.覆蓋深度和GC偏好性
PacBio和Illumina?reads分別覆蓋了P. micrantha葉綠體基因組的100%和99.6%的���,Illumina組裝后含有低覆蓋度的區域(圖2)����,意味著(zhù)7個(gè)contigs只是覆蓋了葉綠體一致性基因組的90.59%(圖1)����;PacBio數據則明顯更為均勻(圖2)��,并且僅僅組裝成一個(gè)contig, 也是形成葉綠體一致性序列的基礎(圖1)����。圖2展示的是PacBio和Illumina在葉綠體基因上的每個(gè)堿基的覆蓋度�,表明PacBio RS的測序結果表現出更加均勻的覆蓋度�����。
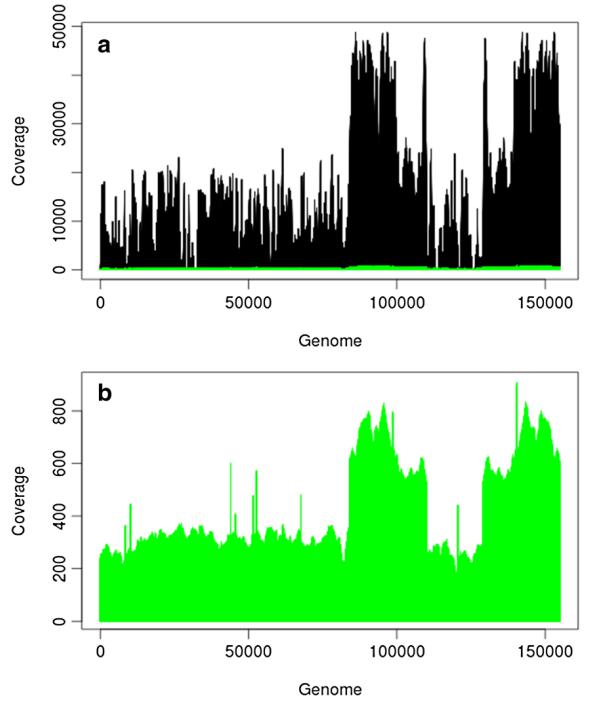
圖2 P. micrantha葉綠體基因組序列每個(gè)堿基的覆蓋范圍?圖中顯示的是葉綠體基因組的每個(gè)堿基之間的測序深度�,(a)表示Illumina (黑色)和PacBio (綠色)測序數據 (b)表示只有PacBio測序數據�,反映出的是PacBio測序數據盡管大大降低了覆蓋度�����,但是在基因組上的覆蓋相對均勻�,對于Illumina測序數據�,還含有很低甚至是0覆蓋度���。此外����,兩個(gè)數據集中顯著(zhù)較高覆蓋度的是反向重復序列����。
為了檢測GC偏性是否存在于這兩類(lèi)測序結果中���,文章對平均覆蓋度和GC百分含量做了相關(guān)性分析��,在計算的過(guò)程中�,排除了反向重復序列���。計算得到的PacBio和Illumina數據集的皮爾森系數分別是0.23 (p-value = 5.675e-09)以及0.61 (p-value = 2.2e-16)��,因此�,Illumina數據集中平均覆蓋度和GC百分含量表現出很強的相關(guān)性(圖3)���。
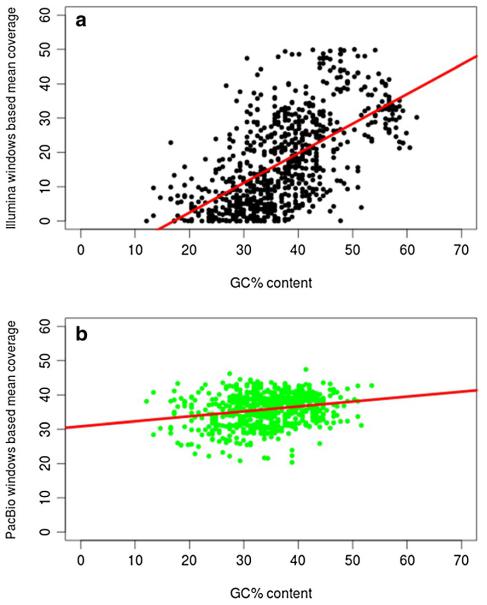
圖3??Illumina and PacBio測序數據集的GC偏性的計算?分別根據(a) Illumina(黑色)(b) PacBio(綠色)測序數據中����,含157個(gè)核苷酸的987個(gè)窗口的平均測序深度�,計算GC含量并作圖�,結果表明�,Illumina數據(皮爾森相關(guān)系數= 0.61�����,p值?= 2.2e-16)比PacBio數據(皮爾森相關(guān)系數= 0.23���,p值?= 5.675e-09)顯示出與GC含量的更強的相關(guān)性���,排除反向重復序列的高覆蓋度數據�����。
?4.錯誤率
組裝前的PacBio RS reads與P. micrantha葉綠體基因組一致性序列相比較�,其錯誤率為1.3%����,而Illumina reads的錯誤率僅是0.117%.
?5.葉綠體基因組針對不同測序深度的組裝
為了了解使用PacBio RS測序數據時(shí)����,測序深度對于P. micrantha基因組組裝的影響�����,使用10×, 20×, 35×, 50×,100×, 150×和200×的樣本數據����,將每個(gè)數據集進(jìn)行組裝����,7個(gè)組裝裝好的數據集中��,有5個(gè)(從?200×到35×)得到的是單個(gè)的contig, 同時(shí)����,測序深度為20× 的數據集得到4個(gè)contigs覆蓋基因組的95.6%���,測序深度為10×的數據集得到的是14個(gè)contigs覆蓋基因組的78.2%. 為了進(jìn)行比較����,Illumina測序數據取與PacBio相同的7個(gè)測序深度的樣本數據���,并進(jìn)行組裝��,但是�����,與9111×測序深度相比����,并沒(méi)有得到比較完整的組裝�。
?6.?P. micrantha葉綠體基因組的結構
組裝好的P. micrantha葉綠體基因組長(cháng)154,959 bp(圖4), 反向重復序列(IR)?25,530 bp, 大的單一重復區(LSC)和小的單一重復區域(SSC)分別長(cháng)85,137 bp和18,762 bp. P. micrantha葉綠體基因組共包含了120個(gè)基因��,有141個(gè)基因功能已知����。其中���,31個(gè)是tRNA編碼基因,7個(gè)位于IR區�����。
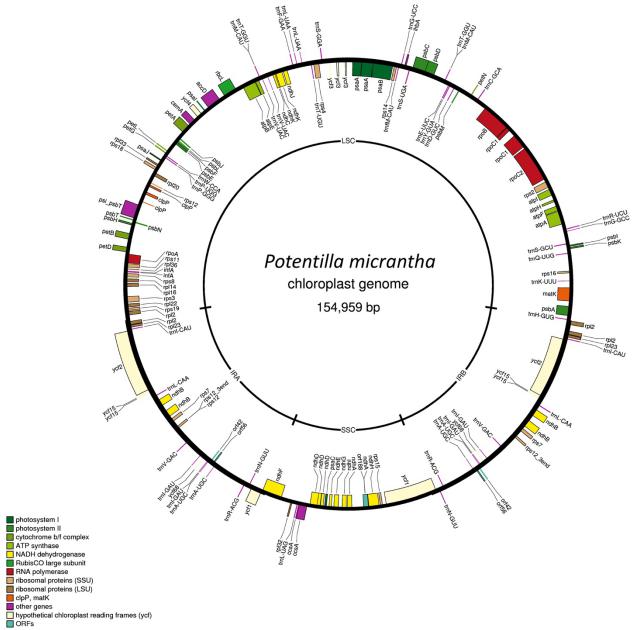
圖4 ?P. micrantha葉綠體基因組序列??P. micrantha葉綠體基因組基因含量的結構圖�����,外圈內側表示基因順時(shí)針轉錄�����,外圈外側表示基因逆時(shí)針轉錄�����,根據不同的功能將基因帶有不同的顏色��;內圈表示基因組的平均GC含量�����。IRa和IRb表示反向重復區域��,LSC和SSC分別表示長(cháng)重復區域和短重復區域��。基因圖譜經(jīng)OGDRAW繪制���。
【研究結論】
1�、 文章第一次報道了使用PacBio RS測序平臺對葉綠體基因組的從頭測序與組裝���,為了更好地評價(jià)PacBio RS測序結果�,文章還將其與Illumina HiSeq2000測序平臺的測序結果進(jìn)行了比較�。
2���、 研究表明���,PacBio測序對于基因組研究具有很大的實(shí)用性���,相比Illumina產(chǎn)生的短reads并且具有GC偏好性低��,它并不會(huì )產(chǎn)生很多gap和contig.
3����、 PacBio RS測序技術(shù)有利于完成不同真核生物高質(zhì)量的測序���。
【所用軟件及數據庫】
SMALT:序列比對工具���,文中用來(lái)過(guò)濾和提取葉綠體DNA reads
AbySS:序列的拼接與組裝是基因組測序數據處理中一個(gè)至關(guān)重要的步驟�����,AbySS是用于高通量測序序列拼接與組裝的軟件
CD-Hit:通過(guò)序列比對聚類(lèi)(Cluster)的方法去除冗除����、相似的序列���,最后輸出一個(gè)非冗除(non-redundant�,nr)的序列文件
SMRT:Pacific Biosciences公司的SMRT技術(shù)�����,作為第三代測序技術(shù)�����,與前兩代技術(shù)相比�����,其最大的特點(diǎn)是單分子測序
DOGMA:基因組注釋工具
OGDRAW:在基因組學(xué)研究中���,用于畫(huà)一個(gè)小的環(huán)形基因組��,例如線(xiàn)粒體��、葉綠體�����、質(zhì)粒的圈圖
CORAL:糾正下一代測序數據錯誤率的工具